Inhaltsverzeichnis
Die Herstellung von Proteinen läuft in den Zellen über viele einzelne Schritte ab. Das Ribosom ist dabei ein außerordentlich wichtiger Bestandteil, denn ohne dieses Molekül würde kein einziges Protein zustande kommen. Dieser Artikel behandelt den Aufbau und die Funktion im Rahmen der Translation im Detail und geht abschließend auf die Bedeutung der Ribosomen bei einer Antibiotikatherapie ein.
Inhaltsverzeichnis
Ribosom – Definition und Vorkommen
Ein Ribosom ist ein großer Molekül-Komplex (Makromolekül-Komplex), welches aus Proteinen und ribosomaler RNA (rRNA) besteht. Es katalysiert die Proteinbiosynthese in Zellen, genau genommen die Translation. Dabei übersetzt es die Information der mRNA in das endgültige Protein.
Ribosomen kommen in allen Zellen vor, die Proteinbiosynthese betreiben. Teilweise finden sie sich frei im Zytoplasma (zytosolische Ribosomen), aber häufig auch am endoplasmatischen Retikulum (membrangebundene Ribosomen). Dieses wird dann als raues endoplasmatisches Retikulum bezeichnet. Reihen sich mehrere Ribosomen entlang der mRNA, um die Translation zu vollziehen, spricht man von einem Polysom.
Eine andere Lokalisation ist im Inneren von Mitochondrien, wo sie der Translation mitochondrialer Proteine dienen.
Ribosom – Aufbau
Allgemein besteht ein Ribosom zu einem Drittel aus Proteinen und zu zwei Dritten aus der ribosomalen RNA (Ribonukleinsäure). Diese Bestandteile verteilen sich auf zwei Untereinheiten des Ribosoms, eine große und eine kleine Untereinheit. Im Elektronenmikroskop erscheinen die Ribosomen rund bis ellipsoid mit einem Durchmesser von etwa 25 Nanometern.
Die Untereinheiten liegen im inaktiven Zustand getrennt voneinander vor. Sie lagern sich unter Einfluss der mRNA, Magnesium und Initiationsfaktoren zusammen, sodass die Translation beginnen kann.
Die Masse der Untereinheiten wird mittels der Sedimentationskonstante in der Einheit Svedberg (S) angegeben. Sie leitet sich von der Sedimentationsgeschwindigkeit ab, die sich bei jedem Teilchen durch die Zentrifugalkraft in der Zentrifuge ermitteln lässt. Wichtig ist zu beachten, dass die Sedimentationskoeffizienten der einzelnen Bestandteile der Untereinheiten nicht einfach addiert werden können, um die Gesamtmasse zu errechnen.
Kleine Untereinheit (40S)
Die kleine Untereinheit besteht aus einer 18S-rRNA und etwa 33 Proteinen. Daraus ergibt sich die Masse von 40S. Diese Angaben beziehen sich allerdings nur auf die Eukaryoten, da die Ribosomen der Prokaryoten anders zusammengesetzt sind.
Die 18S-rRNA entsteht durch die Transkription der RNA-Polymerase I, die zunächst eine 45S-prä-rRNA herstellt. Daraus werden drei Teile geschnitten: die hier wichtige 18S-rRNA, aber auch die 5,8S-rRNA und die 28S-rRNA. Die letzten beiden genannten sind Teil der großen Untereinheit. Die 18S-rRNA ist nicht katalysierend und hat keine codierende Funktion.
Große Untereinheit (60S)
Bei eukaryotischen Zellen setzt sich die große Untereinheit aus rund fünfzig Proteinen und der rRNA zusammen, was die Masse von 60S ergibt. Die rRNA enthält 5S, 28S und 5,8S-rRNA.
Die 28S-rRNA übernimmt katalytische Aufgaben und erhält dadurch die Bezeichnung als Ribozym, da sie ähnliche Funktionen wie Enzyme ausführt. Genau gesagt ist sie eine Peptidyltransferase, das heißt, sie katalysiert die Peptidbindung zwischen den Aminosäuren. Dadurch kann sich erst die Proteinkette bilden.
Die 5S-rRNA wird nicht durch die RNA-Polymerase I, sondern durch die RNA-Polmerase III gebildet. Sie ist die kleinste rRNA.
Auch Prokaryonten besitzen Ribosomen, die sich jedoch im Aufbau von eukaryotischen Ribosomen unterscheiden. Sie bestehen ebenfalls aus zwei Untereinheiten, die kleine setzt sich jedoch aus einer 16S-rRNA und 21 Proteinen zusammen, welche die 30S-Untereinheit bilden. Die große Untereinheit besteht aus der 5S-rRNA und der 23S-rRNA in Verbindung mit 34 Proteinen, woraus sich im Gesamten die 50S-Untereinheit bildet. 50S- und 30S-Untereinheit gemeinsam bilden das funktionale 70S-Ribosom.Ribosomen der Prokaryonten
Ribosom – Funktion
Um die Funktion der Ribosomen zu verstehen, lohnt sich der Blick auf den Ablauf der Translation. Nach erfolgreicher Transkription entsteht im Zellkern die messenger RNA (mRNA), die in das Zytosol oder an das endoplasmatische Retikulum freigesetzt wird. Sie enthält eine Basenabfolge, die im nächsten Schritt in Aminosäuren und damit in Proteine übersetzt wird.
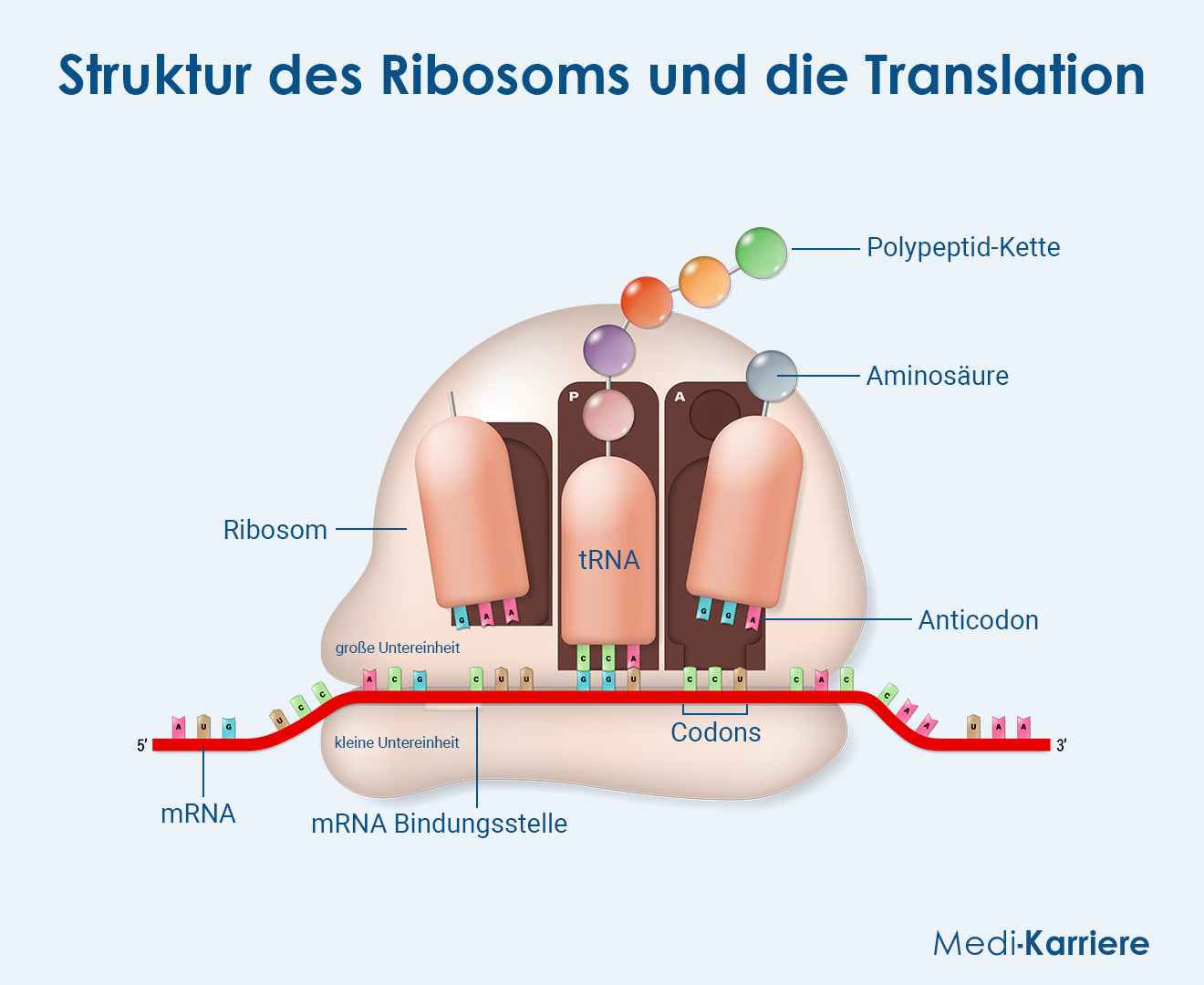
Dazu bindet die mRNA nach Freisetzung zunächst an die kleine Untereinheit, die den Präinitiationskomplex bildet, zu dem noch ein GTP-Molekül, die Initiator-tRNA und weitere Initiationsfaktoren zählen. Die kleine Untereinheit liest die mRNA bis zum Startcodon (Basenabfolge AUG) ab. Ist dieser Punkt erreicht, bindet die große Untereinheit. Sie wird durch die Initiationsfaktoren unterstützt.
Bei der Fusion der beiden Teile entsteht eine Rinne, in der die mRNA entlang gleitet. Außerdem bilden sich folgende drei Bindungsstellen:
- A-Stelle: Aminosäure-Stelle
- P-Stelle: Polypeptid-Stelle
- E-Stelle: Exit-Stelle
An diese binden transfer-RNAs, kurz tRNA. Sie tragen die Aminosäuren und besitzen ein Anticodon, mit dem sie sich an die mRNA anlagern. Das Startcodon codiert für eine tRNA, die die Aminosäure Methionin bindet. Es folgt jetzt ein Verlängerungszyklus, bei dem das Ribosom zwischen zwei Zuständen wechselt: dem prä- und posttranslationalem. Bei beiden sind jeweils zwei der Bindungsstellen von tRNAs besetzt.
Prätranslationaler Zustand
Hierbei sind die A- und P-Stelle besetzt. Die tRNA in der P-Stelle trägt die entstehende Proteinkette, an die A-Stelle werden neue tRNAs mit Aminosäuren angeliefert. Die Polypeptidkette wird durch die große Untereinheit auf die A-Stellen-tRNA übertragen. Hierbei ist vor allem die oben genannte 28S-rRNA als Peptidyltransferase wichtig.
Posttranslationaler Zustand
Ist dieser Vorgang erledigt, rutscht das Ribosom drei Basen, und damit eine Bindungsstelle, weiter. Die vorherige A-Stellen-tRNA findet sich mit der Proteinkette jetzt an der P-Stelle, die vorherige P-Stellen-tRNA sitzt an der E-Stelle. Sie wird im nächsten Schritt freigesetzt. Dieser Vorgang wiederholt sich so lange, bis das Stoppcodon erreicht ist und die Aminosäurenkette abgespalten wird.
Welche Ribosomen translatieren welche Proteine?
Je nach Lokalisation produzieren die Ribosomen Proteine für unterschiedliche Zellbestandteile.
Freie, zytosolische Ribosomen translatieren die Proteine, die im Zytosol vorkommen. Dazu zählt zum Beispiel Tubulin. Die membranständigen Ribosomen an dem rauen endoplasmatischen Retikulum synthetisieren hingegen sekretorische Proteine, die im Anschluss aus der Zelle herausgeschleust werden, und Membranproteine für die Zellmembran.
Das Mitochondrium besitzt eigene Ribosomen, die etwa ein Prozent der mitochondrialen Proteine synthetisieren. Dazu zählen etwa Proteine der Atmungskette.
Ribosom – Klinik
Nicht nur Eukaryoten, sondern auch Prokaryoten wie Bakterien besitzen Ribosomen und brauchen diese zwingend, um weiterzuleben. Pathologische Bakterien können an dieser Stelle mit Antibiotika bekämpft werden.
Makrolide sind eine Klasse, die die bakterielle Proteinsynthese unterbindet, indem sie an die 50S-Untereinheit der Ribosomen bindet. Sie wirken damit bakteriostatisch, was bedeutet, dass sich die Bakterien nicht mehr vermehren und nicht mehr wachsen können. Erythromycin oder Azythromycin sind typische Vertreter dieser Klasse. Ebenfalls an diese Untereinheit binden Lincosamide wie Clindamycin.
An die 30S-Untereinheit binden Vertreter der Aminoglykoside, die dadurch die gleiche Wirkung erzählen. Gentamicin und Streptomycin sind Beispiele hierfür.
- Löffler/Petrides et. al., Biochemie und Pathobiochemie, 10. Auflage, Springer
- Aufbau von DNA und RNA, https://next.amboss.com/... , (Abrufdatum: 27.07.2024)
- Die Zelle, https://next.amboss.com/... , (Abrufdatum: 27.07.2024)
- Antibiotika – Übersicht, https://next.amboss.com/... , (Abrufdatum: 27.07.2024)